ニュース
DNA分子モーターの動きをナノスケールでコントロールする事に成功【京都大学】
2012年1月23日
科学技術振興機構(JST)課題達成型基礎研究の一環として、京都大学(総長:松本紘)と英国オックスフォード大学(総長:クリス・パッテン)は、約100nmのDNA平面構造上に作成した経路で、DNAで作成した分子モーターの進行をナノスケールの精度で人為的にコントロールする技術を世界で初めて実現しました。これらの人為的な分子の運動の操作は、狙った所への分子の運搬や分子ロボットの基礎に役立つ技術となります。
杉山弘 物質-細胞統合システム拠点(iCeMS=アイセムス)・理学研究科教授、遠藤政幸 iCeMS准教授らの研究グループは、DNA オリガミと呼ばれる1辺100nm程度の長方形平面構造体に進行方向のガイドとなるDNAでできた「レール」が複数分岐したものを作成し、そのレールに沿ってDNA分子モーターを思った位置に移動させることに成功しました。さらに分岐点で、進行方向を人為的にコントロールし、ナノスケール下での分子の動きを制御し、高速原子間力顕微鏡(AFM)を使って数ナノメートルの解像度で観察することに成功しました。本成果により、あらかじめ設計したナノ・メゾ空間での分子の動作のコントロールが可能となり、ナノ・メゾ空間での分子の輸送や分子ロボットの開発につながるものと期待されます。
本研究は、JST戦略的創造研究推進事業チーム型研究(CREST)「プロセスインテグレーションに向けた高機能ナノ構造体の創出」研究領域における研究課題「生体分子情報-構造-機能統合ナノシステムの構築」(研究代表者:杉山弘)の一環として行われました。この成果は、ロンドン時間2012年1月22日午後4時(日本時間23日午前3時)に英科学誌「ネイチャー・ナノテクノロジー」オンライン版で公開されました。
1. 背景
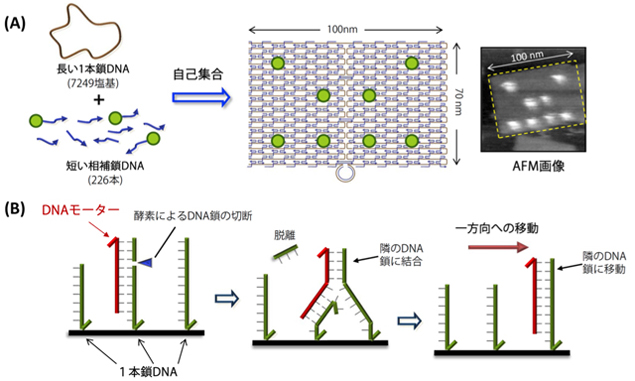
分子の大きさの世界であるナノメートルスケール(1nmは10億分の1m)で、分子を思ったとおりに並べて動かすテクノロジーは、化学の分野だけでなく、生物や物理など科学全般に重要なテーマです。分子は生物を構成する最も小さな単位であり、その分子が遺伝情報などのプログラムに従って規則的に集合し、組織化されることで高度に機能する生物に組みあがっていきます。こうした、規則に従って分子が集合する現象である自己集合を人為的に行うためには、あらかじめ分子に指令(プログラム)を書いておき、その指令に従って集合させる必要があります。DNAは四つの塩基がテープ状に配列された分子であり、塩基配列というプログラムにしたがって、2重らせんの形成を行います。同研究グループは、DNAの配列を設計することでさまざまな構造体を作成して、その上にさらに分子を思ったとおりに並べる技術を開発しています。この技術はDNAオリガミ法と呼ばれます(図1Aおよび用語解説参照)。この技術を使えば、一つ一つの分子を作成した構造上の好きな場所にナノメートル単位で正確に置くことができます(図1A)。分子を見る装置は原子間力顕微鏡(AFM)で、数ナノメートルの大きさの分子を見分けることができます。同研究グループでは、分子モーターが動く様子をAFMで直接観察する方法も開発してきました。さらにこれを応用して、複雑な経路をDNAオリガミ上に作成し、あらかじめ組み込んだプログラムに従って、DNAモーターの進行方向をナノスケールでコントロールできる手法を開発し、この動きをAFMで捉えることに挑戦しました。
2. 研究内容と成果
ナノメートルの世界で機械的な動きを再現できる分子機械は、反復運動や回転運動、移動など様々な運動ができるものが開発されています。今回の研究で用いたのはDNAを使って作成したDNA分子モーターと呼ばれるもので、1方向へ移動していきます(図1B)。この中で、1列に並んだ1本鎖DNA(図1B 緑色のDNA)に対してその相補鎖DNA(DNAモーター、図1B 赤色のDNA)が結合し、2本鎖DNAとなると、この塩基配列で酵素が切断し、1本鎖DNAが短くなります。これによって、DNAモーターはより安定に結合できる隣の長い1本鎖DNAへ移動していきます。この反応が繰り返されれば、DNAモーターは1列に並べた1本鎖DNA に沿って順次移動していくことになります。次に、このDNAモーターが動くためのレールとなる部分を作る必要があります。これには数ナノメートルの精密さで分子を決められた場所に並べる技術が必要になります。ここでは、DNAオリガミを使って平面構造を作り、その上に設計したとおりのDNAモーターが動く1本鎖DNAからなる経路(レール)を作成しました(図2)。今回の研究では、経路が複数分岐したところを作り、指令に従って、DNAモーターの進行方向がコントロールできるか試みました。
図1. (A) DNA オリガミ法を用いたナノメートルスケールの構造体の作成方法。短い相補鎖DNAに目的とする分子(緑丸)を結合しておけば、DNAオリガミ構造体上の好きな位置に分子を配置できる。分子を配列したDNA構造体のAFM 画像。 (B) DNAモーターの動作原理。酵素反応によって、固定した1本鎖DNA(緑色)を介してDNAモーター(赤色)は1方向へ移動する。
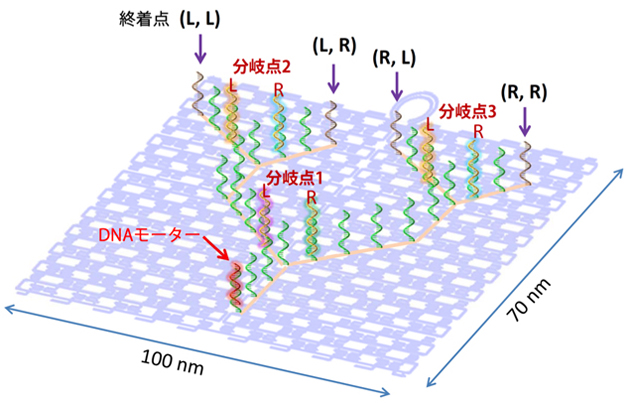
図2. DNA オリガミ構造上への1本鎖DNAによる経路(レール)の導入。経路には三つ分岐点とその両側にゲート(LまたはR)を作った。終着点は4箇所。図は酵素反応前の初期構造。
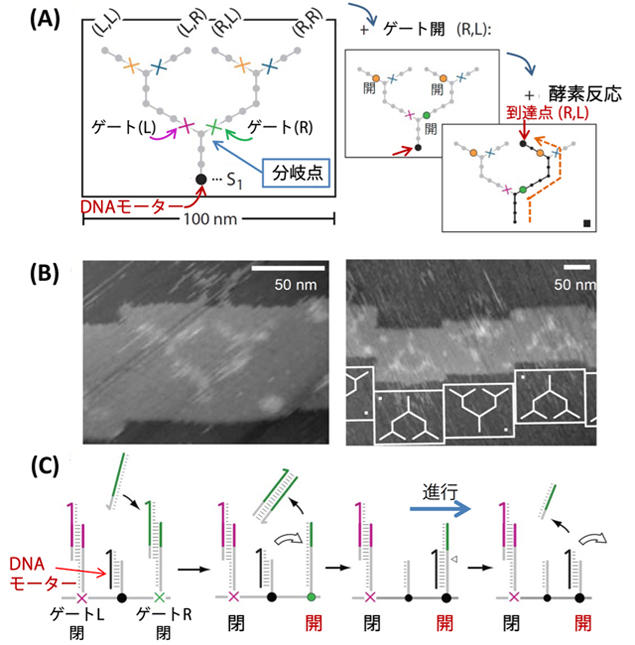
DNAオリガミ法で作成した長方形の構造体の上に、1本鎖のDNAを等間隔に並べ、三つの分岐点がある経路を作成します( 図2 および図3A)。AFM画像では作成した経路の形をその通りに見ることができます(図3B)。この経路のスタートの位置にDNAモーターを配置します。酵素反応により、DNAモーターはこの位置から順次隣のDNA鎖に移動していきます。まず、最初の分岐点でDNAモーターは右か左かのどちらかに移動します。ここに、DNAモーターの進行を妨げる分子を導入しておけば、そちらの方向へは進めなくなります。つまり、ゲートの役割を果たすことができます。ゲートの仕組みは図3Cのようになり、あらかじめ、両側のゲートに2本鎖を形成させておけば、DNAモーターはどちらにも通過することができなくなります。このゲートの片側を開く、つまり、ゲートを形成している2本鎖から相補鎖を引き抜けば、そちらを通過できることになります。これにはゲートの相補鎖に余分な配列をつけておき、それと完全に相補的なDNAを加えて結合させれば、引き抜くことが可能です。そうすれば、開いた方は1本鎖となり、そちらにDNAモーターが進行できるようになります(図3C)。このようにあらかじめ設計した配列のDNAでゲートを開くことができ進行方向をコントロールできます。
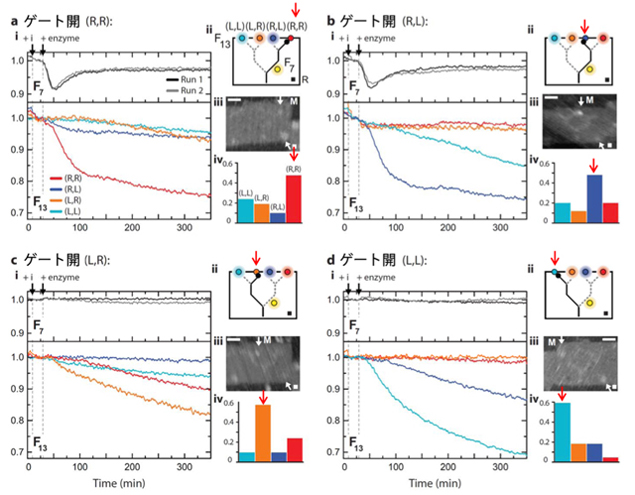
ここでは、三つの分岐点の左右両側にゲートを設け、DNAモーターは二つの分岐点を通過して、最終的に四つの終着点までたどり着き、そこで停止します。1番目と2番目の分岐点でゲートを両方とも左に開いておけば、DNAモーターは最も左の終着点(L, L)の位置に到達します。同様に1番目の右のゲートと3番目の左のゲートを開いておけば、終着点(R, L)の位置にDNAモーターを誘導できます(図3A)。このDNAモーターの動きをAFMで時間を追って観察していきます。DNAモーターにはビオチンと呼ばれる分子が結合してあり、酵素反応後にタンパクであるストレプトアビジンをビオチンに結合することで、DNAモーターの位置をAFMによって観察します(図4)。DNAモーターが最終的に到達した位置を調べると、ゲートの開いた方向へ、DNAモーターが進行していくことが明らかとなりました。つまり、分岐点でのゲートの開閉によって、DNAモーター分子の進行をコントロールでき、最終地点への誘導が可能であることが確かめられました。また、水溶液中でもこのDNAモーターの進行方向のコントロールを確認しました。DNAオリガミ上の終着点に異なる蛍光基をあらかじめ配置しておき、DNAモーターに消光基(接近したときに蛍光が出ないようにする分子)を結合し、同じようにゲート操作とDNAモーターの進行を行うと、ゲートの開いている経路の最終地点の蛍光だけが弱くなる(消光される)ことがわかりました(図4)。つまり、この系はAFM測定のような表面に固定した1分子観察だけでなく、水溶液中でも同様に進行することが確認できました。
このように、本研究ではDNAで作成した分子モーターをDNAオリガミで作成した構造上に作成した経路でその進行をナノスケールの精度で人為的にコントロールする方法を世界で初めて実現しました。
図3. (A)DNA構造体に作成した1本鎖DNAからなる経路と(B)そのAFM画像。(C)分岐点の両側に作成したゲート(Lが左側のゲートでRが右側のゲート)とそれを開く仕組み。
図4. DNA構造体の経路上での酵素反応後のDNAモーターの動き。4種類のゲート操作(a-d)とそれに対応する蛍光強度の時間変化とAFM画像及びDNAモーターの位置の分布。AFM画像上の矢印の先の白いスポットがDNAモーター(Mで表示)。
3. 今後の期待
今回の研究では、DNAオリガミ上にナノスケールのレールを敷き、複雑に分岐した経路と進行方向を決められるゲートを使って、分子モーターを思った通りの位置に動かす技術を開発しました。ナノスケールの精密さで分子の移動をコントロールする技術は開発段階であり、まだまだ展開していく可能性を秘めています。ここで示したとおり、ナノメートル単位で分子を思ったとおりに並べ操る技術や分子の動きをコントロールする技術はナノテクノロジーの重要課題であります。この研究のように思った通りに分子を組み上げ、DNAの配列、つまりプログラムに応じた分子の動きの操作が可能となり、ナノ・メゾ空間での輸送型の分子ロボットなどの開発につながるものと期待されます。
用語解説・注釈
DNAオリガミ
DNAから自己集合によって作成されるナノ構造体。2006年にカリフォルニア工科大学のPaul Rothemund(ポール・ロスムンド)博士によって開発された。鋳型となる長い1本鎖DNA(7,249塩基)に設計した短い相補鎖DNAを加え、加熱、ゆっくり冷却することで、あらかじめ設計したDNAナノ構造体と同じ形に自己集合させることができる。この方法で、長方形、三角形、星型、スマイルマークなどを作成することが可能である。
原子間力顕微鏡(AFM)
走査プローブ顕微鏡の一つ。試料と鋭い探針の間に働く原子間力を利用して、試料表面の形状を測定する。探針はカンチレバー(板状のバネ)の先端に取り付けてあり、試料との間に原子間力が働くとカンチレバーがたわむので、その変位をカンチレバー背面へのレーザー照射とその反射光の検出によって測定する。試料台に取り付けられた圧電素子の制御によって試料をナノメートルスケールで走査することが可能である。今回の実験で使用したAFMは1秒間に15画像を取り込むことができる高速AFMである。
メゾ
「中間」を意味し、主に数ナノメートルから数百ナノメートルの領域を指す。分子は大きくても数ナノメートルであり、一方で細胞などの機能はマイクロメートルで発現する。その中間に存在するメゾスコピック領域は、生命現象の理解や結晶中における分子の「協同的な」動きといった、様々な課題において重要となる。メゾ科学は、幹細胞(ES/iPS細胞など)研究と並び、iCeMSのキーコンセプトである。
ネイチャー・ナノテクノロジー(Nature Nanotechnology)
ナノテクノロジーに関する全ての領域において、特に秀逸で影響力の高い論文を掲載する学際的ジャーナル。化学・物理学・物質
科学・バイオメディカル・工学といった、幅広い分野間の学際的な研究を奨励する。
関連リンク
●iCeMSウェブページ
http://www.icems.kyoto-u.ac.jp/j/pr/2012/01/23-nr.html
● 論文は以下に掲載されております。
http://dx.doi.org/10.1038/NNANO.2011.253
http://hdl.handle.net/2433/152384 (京都大学学術情報リポジトリ(KURENAI))
書誌情報
“A DNA-based molecular motor that can navigate anetwork of tracks”
Shelley F. J. Wickham, Jonathan Bath, YousukeKatsuda, Masayuki Endo, Kumi Hidaka, Hiroshi Sugiyama, Andrew J. Turberfield
Nature Nanotechnology, DOI: 10.1038/NNANO.2011.253
京都大学ホームページはこちら